Cas12a属于II类V-A CRISPR系统,它是用CRISPR RNA (crRNAs)编程,通过RuvC结构域特异性识别和切割dsDNA或单链DNA (ssDNA)。Cas12a在识别其目标dsDNA时,可以释放单链脱氧核糖核酸酶(ssDNase)活性,不加区分地切割附近的ssDNA(即反式切割)。基于 CRISPR/Cas12a 系统的传感方法在恒温条件下就可以进行,不需要很高的温度,在一定程度上简化了核酸的检测过程。
研究者已经将其广泛应用于核酸物质和非核酸物质的检测,也进一步应用到了临床样本的检测,相比于其他的方法展现了很强的优越性。但是,尽管 CRISPR/Cas12a 存在众多优点,也不可避免的存在一些缺陷,比如很多报道的关于CRISPR/Cas12a系统的传感器都严格要求目标DNA含有 TTTN 的 PAM 位点,这就大大地限制了 CRISPR/Cas12a 在核酸分析中的广泛应用。此外,每检测一种新的核酸序列都需要设计一种相应的特异性 crRNA,而 crRNA 的序列设计又相对复杂和繁琐,且合成成本也比较高。因此,建立一种不依赖于含有PAM序列的目标核酸,并且不需要多次设计crRNA的通用的核酸检测新方法尤为重要。
近期,我校夏乾峰教授研究团队在国际期刊《Analytica Chimica Acta》(中科院分区二区,IF:5.977)上发表了论文题目为《A universal CRISPR/Cas12a nucleic acid sensing platform based on proximity extension and transcription-unleashed self-supply crRNA》,夏乾峰教授以及重庆医科大学附属第一医院临床分子医学检测中心技术负责人程伟教授为共同通讯作者,海南医学院2018级硕士研究生田国振为第一作者。
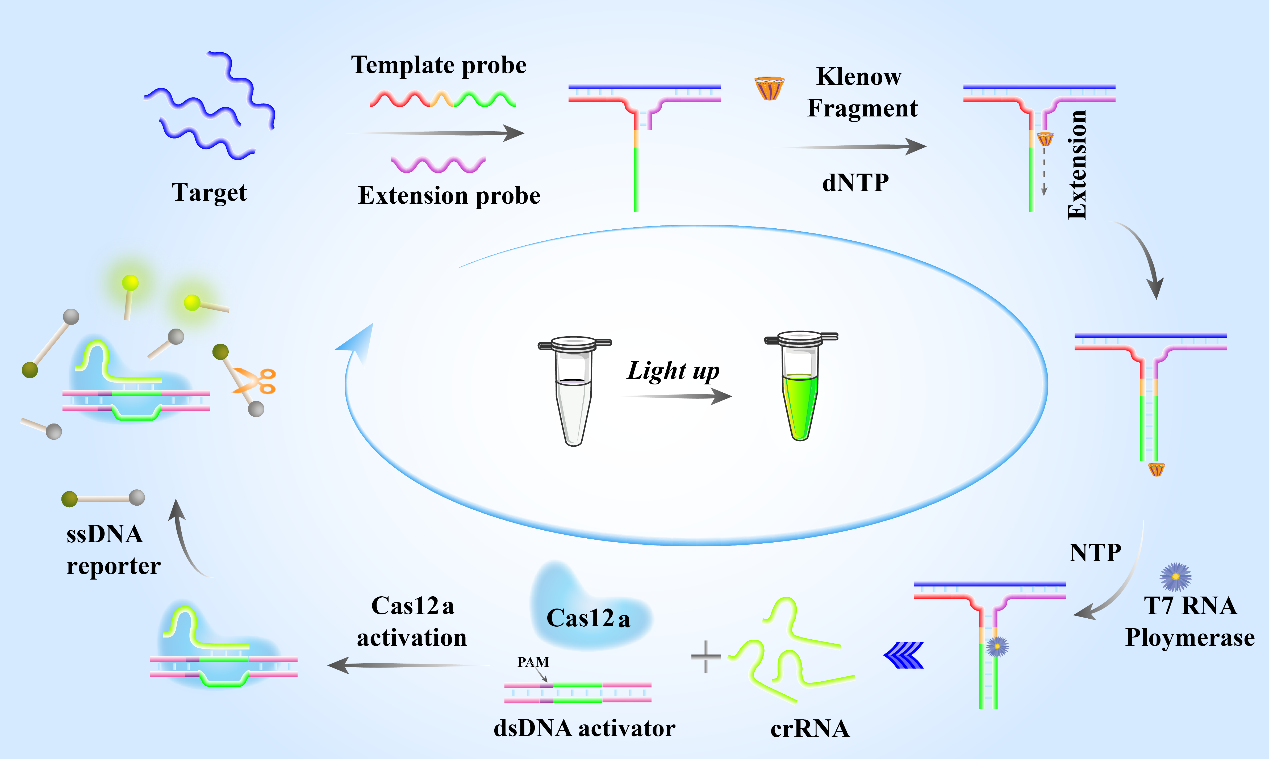
该研究在自然科学基金、海南省科技重大专项、国家科技重大专项和重庆市杰出青年科学基金的资助下完成。研究通过模板探针和延伸探针来捕获靶核酸,激活延伸和转录扩增反应,进而激活Cas12a的反式切割活性,切割ssDNA荧光探针。由于邻近识别的独特设计,基于CRISPR的传感策略可以用于广泛的DNA和RNA序列的一般检测。此外,由于crRNA以高度特异性的目标响应方式自发地从该系统转录,可以显著消除背景,从而提高检测的准确性。同时,这种策略简化了crRNA的设计,降低了检测成本。该方法简单、稳定、经济、灵敏、特异,为CRISPR/Cas12a在临床核酸检测方面的进一步发展奠定了基础。
论文链接:https://doi.org/10.1016/j.aca.2021.338755